背景:性染色体非整倍体疾病(Sex Chromosome Aneuploidies,SCAs)、21-三体综合征、18-三体综合征和13-三体综合征是临床上常见的人类染色体疾病。但是目前的NIPT仅集中于21-三体综合征、18-三体综合征和13-三体综合征的检测,对性染色体非整倍体的检测灵敏度和特异性显著低于21-三体综合征、18-三体综合征和13-三体综合征。有研究表明,限制性胎盘嵌合、胎盘嵌合和胎儿嵌合是导致SCAs检测准确性降低的原因。我们推断,母体染色体嵌合也是造成NIPT性染色体非整倍体检测不符的另一重要原因。
方法:我们建立了一种基于大规模平行测序技术检测染色体嵌合比例的快速染色体核型分析方法,该方法经XXX和XO混合的DNA嵌合体模型的模拟实验验证,并应用于NIPT SCA检测不符的母体白细胞(WBC)DNA检测。
结果: 染色体测序核型分析方法可检测低至5%的X染色体嵌合体,实现了母体X染色体核型的准确检测。在一项NIPT的前瞻性研究中,我们发现181例SCAs阳性患者中有16例(8.6%)是由于母体X染色体核型异常而掩盖了胎儿X染色体DNA片段的真实情况。
结论:通过结合母体血浆测序和母体白细胞测序的结果,可大大提高NIPT对性染色体非整倍体的检测准确性。鉴于母体嵌合发生的高概率,对于NIPT(基于鸟枪法或单核苷酸多态性)所发现的胎儿SCA,都必须进行母体白细胞检测。
染色体非整倍体是生殖细胞减数分裂或着床前受精卵有丝分裂出现错误导致(1)。绝大多数非整倍体疾病是致命的,会引起胚胎着床前停止发育或怀孕前三个月胎儿自发流产。常染色体三体、多倍体以及X染色体单体是导致早期妊娠失败的主要染色体畸变(2-4)。小部分的染色体非整倍体疾病如21-三体综合征(唐氏综合征)、18-三体综合征(爱德华氏综合征)、13-三体综合征(帕陶氏综合征)、X染色体单体(特纳综合征)、XXY综合征(克氏综合征)、XXX综合征(三X染色体综合征)和XYY综合征(超雄综合征),在胎儿发育的中孕期和晚孕期不会引起妊娠失败(原因不明),从而导致0.3%的染色体疾病患儿出生(5)。30多年来,产前诊断的临床应用,加强了针对胎儿染色体非整倍体的检查,减少了染色体疾病患儿的出生(6-8)。母体血清学筛查技术、超声检查、绒毛膜取样或羊水穿刺并结合胎儿核型分析,对常染色体21、18、13三体综合征有较高的准确性(6,9)。相反,除X染色体单体外,由于大多数胎儿性染色体非整倍体(SCAs)或SCA嵌合体无明显临床症状或超声异常,仍然无法精确诊断。
随着母体血浆大规模平行测序的临床应用,孕妇可选择无创产前检测(NIPT)对21、18和13三体综合征以及SCAs进行产前检测,这些染色体非整倍体疾病大约占胎儿染色体畸变的一半(12)。前瞻性临床研究结果显示,NIPT对T21、T18和T13具有很高的检测灵敏度和特异性,均达100%(13-15)。虽然也存在假阳性和假阴性案例,但发生概率极低(<0.1%)。对有限的NIPT检测不符案例进行进一步的胎儿核型分析与胎盘细胞遗传学分析研究发现,胎盘限制性嵌合(CPM)(16-18)和胎盘嵌合(18-21)是导致NIPT检测不符的重要原因。胎盘限制性嵌合和胎盘嵌合可增加胎儿有效DNA片段浓度导致假阳性结果,也可降低胎儿有效DNA片段浓度导致假阴性结果。而对于SCAs,尽管NIPT也具有较高的检测精确度,但在已知染色体核型的回顾性双盲研究(22、23)和前瞻性研究(13、15)中均报道,NIPT对SCAs的检测灵敏度和特异性较低。尽管通过改进对同源染色体X和Y测序所得序列的定位方法,已提高了X染色体和Y染色体Z值计算的精确度,但研究人员仍普遍认为绝大多数 SCAs NIPT检测结果不符是由CPM、胎盘嵌合、胎儿嵌合引起的(9)。
导致SCAs NIPT检测不符的另一个可能原因是母体染色体异常或母体染色体嵌合。因为母体染色体异常或母体染色体嵌合,母血浆中X染色体的有效DNA片段与核型正常的孕妇不同,进而会影响母血浆中真实胎儿X染色体DNA片段的计算。我们最近报道了1例因母体 6% T18嵌合造成的NIPT T18假阳性案例(15),从原理上证明了母体染色体异常可显著影响胎儿相应染色体的Z值计算。另外1例13号染色体三体和18号染色体单体的NIPT假阳性案例,经临床随访发现是由于母亲患有转移性肿瘤(25)而引起。不过也有小部分健康、具有生育能力的SCA嵌合或XO或XXX孕妇可生育染色体正常的后代(11)。另外也有报道,一些高龄孕妇的X染色体会逐渐缺失导致X染色体失活,从而使得核型由XX变为XO/XX嵌合(26)。有一项单独的研究发现低水平的母体45,XO嵌合可导致NIPT检测结果不符(18)。因此我们推断,孕妇染色体核型异常会影响其血浆中X染色体DNA片段的浓度,在接受NIPT检测时可能会出现胎儿SCAs NIPT检测不符的情况,这种案例虽然很少,但非常值得关注。本研究旨在建立一种快速、准确检测母体SCAs的方法,并确定母体染色体异常导致胎儿SCAs NIPT检测不符的发生概率。
方法
DNA样品制备与核型分析
参与本研究的孕妇为12-16孕周的中国孕妇,我们对每位孕妇采用Cell-free DNA BCT?管(美国Streck公司)采集10ml外周血,并将血液样本运输至北京贝瑞和康生物技术有限公司进行样本处理和测序。取2.3ml外周血,经2次离心后分别得到约1.3ml血浆和1.0ml白细胞(WBCs)。分别提取血浆和白细胞的DNA:取1.0ml血浆,使用QIAamp Circulation Nucleic Acid Kit试剂盒(德国Qiagen公司)提取其DNA。取0.2ml母体白细胞提取其基因组DNA。为了更好的分析检测灵敏度和重复性,我们按照上述方法采集2ml健康男性(46,XY)和X三体综合征病人(47,XXX)的外周血作为对照样本。通过中期细胞450条带的染色体G显带技术对羊水细胞或母体白细胞进行核型分析。
无创DNA产前检测
参照已发表研究中的实验方法,首先构建血浆DNA文库,然后采用Illumina HiSeq 2000 平台进行大规模平行测序(15,27)。每个测序文库产生大约8M的36bp单端测序数据,将这些数据与人类参考基因组进行比对(28)。统计与人类参考基因组唯一匹配的序列数,按照GC矫正的方法,分别计算21号、18号、13号以及X染色体的Z值(27)。对于Z值小于-3.0或大于+3.0的,判为染色体异常(15)。
母体白细胞基因组测序检测母体核型异常
对于每个样品,取50ng白细胞基因组DNA,采用Nextera DNA 样品制备试剂盒(Epicentre公司/Illumina公司)构建测序文库。多个测序文库index标记后混合至HiSeq 2000测序仪的单一通道,进行43bp单端测序(其中7bp为index序列)。对测得的序列片段按图1进行数据分析。首先,将测得的序列与已知人类参考基因组比对(hg19)。然后,通过将每条染色体分为连续的20kb bins计算序列密度。对于特定样品的每个 bin i,统计这个bin中唯一并完美匹配的序列数(RNBi),然后利用这个样品测序所得的序列总数和这个bin i中含有的所有可能的唯一36bp序列数(Ui)进行标准化处理,计算公式如下:NRNBi=RNBi/(样品序列总数)×(8×106)/Ui,该公式中的NRNBi指与bin i唯一并完美匹配的标准序列数。对于对照样品的任何bin i,则计算NRNBi的中值(μi)。我们假设对于每个bin,对照样品之间异常拷贝数鲜有变化,因此,NRNBi的中值μi 可代表染色体正常拷贝数。对于待测样品的每个bin i,我们计算了待测样品标准序列数和正常拷贝数的比值(ratioi),计算公式如下:ratioi= NRNBi/μi。然后我们标绘了待测样品所有bins的log2(ratioi)值,以计算出每条染色体的拷贝数值。参照已发表研究中的Fused Lasso算法确定染色体片段的重复或缺失(29)。最后,参照已发表研究中的方法分别计算每个待测样品j的每条染色体的归一化染色体代表值(NCRj )和参考样品f的每条染色体的归一化染色体代表值(NCRf),以确定染色体的嵌合比例(27)。染色体的嵌合比例计算公式如下:(NCRj—NCRf)/ NCRf。
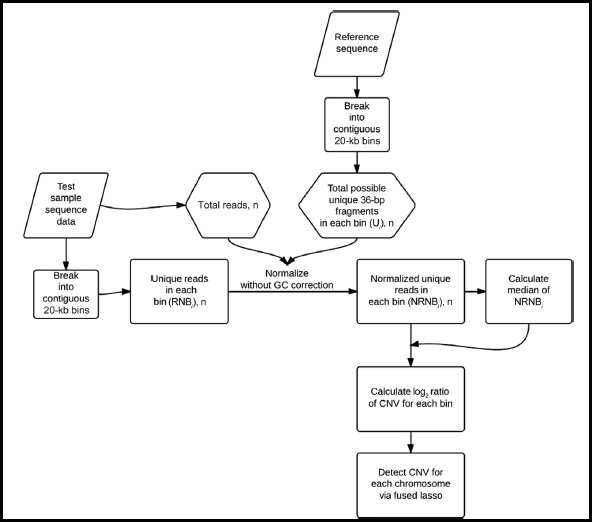
Reference sequence | 参考序列 |
Break into contiguous 20-kb bins | 分为连续的20kb bins |
Test sample sequence data | 待测样品序列数据 |
Total reads, n | 测得的序列总数 |
Total possible unique 36-bp fragments in each bin (U),n | 所有bin 中含有的唯一36bp片段数(Ui) |
Unique reads in each bin (RNBi), n | 每个bin的单一序列数(RNBi) |
Normalize without GC correction | 无GC校正的标准化 |
Normalized unique reads in each bin (NRNBi), n | 标准化每个bin(NRNBi)的单一序列 |
Calculate median if NRNBi | 计算NRNBi中值 |
Calculate log2 ratio of CNV for each bin | 计算每个bin的CNV log2 ratio |
Detect CNV for each chromosome via fused lasso | fused lasso法确定每条染色体的CNV |
图1 母体染色体核型异常测序数据分析流程图
CNV,拷贝数变异
结果
母体染色体核型异常有效测序方法的建立和验证
为确定母体嵌合水平对胎儿SCAs NIPT检测结果的影响,我们认为有必要建立一种快速、准确的方法。由于母体白细胞被认为是母体血浆DNA稳定片段的最主要来源,我们认为结合母体白细胞和血浆DNA的分析可获得最准确的检测结果(9)。我们假设将基于DNA测序的NIPT方法同样应用于母体白细胞,可快速鉴定出母体染色体核型异常和/或母体性染色体不同水平的嵌合。为验证此假设,我们利用从47,XXX患者和46,XY正常男性提取的基因组DNA建立了不同母体X染色体嵌合水平的模拟试验。在本试验中,我们以不同比例混合已知的46,XY和47,XXX基因组DNA,从单纯的46,XY (等同于45,XO)DNA开始,然后逐渐增加47,XXX DNA的比例(分别增至5%、10%、20%、30%、40%、50%、60%、70%、80%、90%、95%),以制备XO比例逐渐减少的XO嵌合、XX均衡核型(X染色体无变化)、以及XXX比例逐渐增加直至单纯XXX核型的梯度模拟样品。我们对这些样品进行测序,并计算了X染色体拷贝数的增加比例。
XO(100%)、XO(80%)/XXX(20%)、XO (50%)/XXX(50%)、 XO (20%)/XXX (80%)和XXX (100%)样品的X染色体测序结果如图2所示。测序得到的所有XO嵌合比例和XXX嵌合比例均与预期数值相同。为评估测序方法的重复性,我们针对每种XO和XXX嵌合比例进行了3次独立重复试验(分别由3名技术人员进行)。根据3次独立重复试验的结果,随着X染色体比例的增加,我们描绘了测序所得的X染色体XO/XXX比值(试验值)和预期XO/XXX比值(理论值)之间的线性关系图(r2=0.99893),如图3所示。更为重要的是,我们可以精确检测出低至5%的XO嵌合,可见我们的“测序核型分析”法具有很高的灵敏度。如预期一样,X染色体嵌合DNA样品中的所有常染色体的平均log2值未偏离0,表示常染色体拷贝数正常。
白细胞检测确定NIPT SCAs检测结果不符的母体核型
按常规NIPT检测,我们鉴定了3例X染色体Z值异常高的样品(>5),3例X染色体Z值异常低的样品(<-5),但不能仅从统计学上判为胎儿SCA(表1)。我们推测X染色体Z值异常是由于异常的母体白细胞核型影响了母血浆中X染色体有效片段的计算。为进一步验证我们的推测,我们采用新开发的测序核型分析方法对NIPT血液样品中的白细胞进行了检测。其中6例母体白细胞样品中的4例X染色体核型分析测序结果如图4所示。根据X染色体的增加比例(表1),Z值异常高的NIPT样品1、4、6的母体核型分别为100% XXX、 48% XXX 嵌合和 94% XXX嵌合。与此相对应,根据X染色体的缺失比例,Z值异常低的样品2、3、5的母体核型分别为5%XO嵌合、20%XO嵌合和 15%XO 嵌合。在6个样品中发现,胎儿X染色体DNA片段的变化量与母体WBC DNA的X染色体变化量普遍相关。考虑到胎儿DNA仅占母血浆DNA的一小部分,我们认为母体核型异常是影响胎儿X染色体Z值的主要因素(表1)。为了确认和验证母体核型测序方法的准确性,我们也通过G显带技术对母体WBCs进行了核型分析(表1)。除样品2外,G显带技术得出的核型与测序得到的核型高度一致。进一步行羊膜穿刺G显带核型分析结果显示,6名胎儿均为染色体二倍体(表1)
母体嵌合对NIPT SCAs检测不符的影响
为确定母体嵌合对NIPT SCAs检测不符的影响,我们调查了从2012年12月至2013年4月5个月内NIPT临床检测出的SCAs案例,在这期间NIPT仅出具21、18和13三体综合征的检测报告。调查中共发现446例NIPT结果异常案例,其中259例(58.1%)为21、18或13三体综合征;187例(41.9%)为SCAs。对187例NIPT SCAs检测结果阳性的案例(包括上述6例)进行母体核型测序分析,结果显示,16例(8.56%)母体核型异常或嵌合,其中6例(6/63,9.52%)发现母体X染色体增加,10例(10/124,8.06%)发现母体X染色体减少(表2)。对于剩下的259例胎儿三体综合征案例,母体染色体测序结果均为正常的46,XX核型。
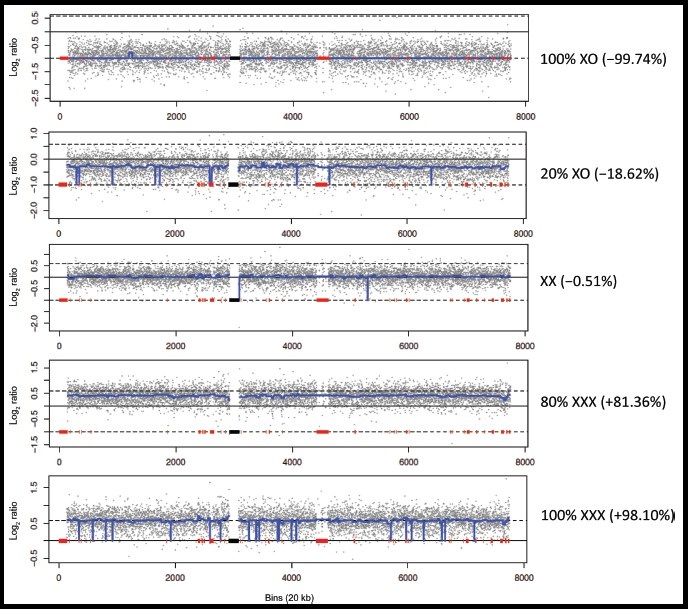
图2. X染色体嵌合模型的核型测序分析
X染色体测序结果图,以标准化测序密度的log2值为Y轴,以20kb测序bin数为X轴。上虚线表示100%X染色体增加[log2(3/2)]。下虚线表示100% X染色体减少[log2(1/2)]。蓝色线代表样本log2值的平均值,红色方框表示序列重复区域,黑色方框表示着丝粒。100% XO、20% XO嵌合、XX、80% XXX嵌合和100% XXX的模型均标注了测序法测定的X染色体的实际平均含量变化(见括号)。
讨论
对NIPT检测21三体综合征(17,20,21)、18三体综合征(20)和13三体综合征(19)不符案例的随访和胎盘分析结果显示,限制性胎盘嵌合、胎盘嵌合是造成上述不符的重要原因,(9)也被认为可导致NIPT SCAs检测不符。在该研究中,我们采用了一种基于测序的、具有高灵敏度和特异性的母体白细胞检测方法,对NIPT性染色体检测异常的187例案例进行了分析,并确定了16例是由于母体染色体异常或嵌合而引起。此种异常会改变母血浆中异常X染色体的含量,是导致NIPTSCAs检测不符的一项重要原因。本研究首次部分诠释了NIPT SCAs检测灵敏度和特异性较低的问题(13,15)。但进一步的原因阐释需要对大量NIPT SCA异常样本进行随访研究,这是目前做不到的。
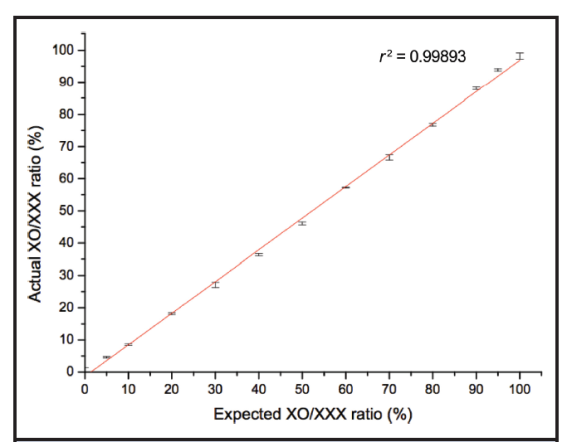
图3:测序方法检测X染色体嵌合的灵敏度和重复性
采用47,XXX 和46,XY的基因组DNA构建不同比例的XO/XX嵌合体和XX/XXX嵌合体模型。根据三次重复试验的平均值+3 SDs,对试验的XO/XXX比率(试验值)和预期的XO/XXX比率(理论值)作图。随着X染色体的增加,不同模型的测序结果之间存在线性关系(r2 = 0.99893)。50% XO/XXX的比率相当于XX,代表着从XO到XXX嵌合的过渡点。
确定母体染色体核型对提高NIPT性染色体检测结果准确性的重要性至关重要。针对此目的,我们专门研发并验证了一种快速染色体测序核型分析方法。这种方法检测的是从孕妇外周血中分离的母体白细胞,孕妇外周血也可用于分离血浆DNA进行NIPT检测。在我们研究的6例NIPT SCAs 检测不符的案例中,5例通过测序方法和G显带核型分析检测到了相同的X染色体缺失或重复。剩余的1例样本(表1中的样本2),G显带核型分析比测序方法检测出较高的45,XO嵌合。鉴于X染色体 NIPT检测的-7.95的Z值更符合测序所预测的相对较小的X染色体缺失,我们认为该样本G显带和测序检测结果不符很可能是由其它因素引起,而这些因素与G显带核型分析相关,比如:未达最佳标准的培养条件、45,XO在扩大培养中发生假性镶嵌混合甚至是细胞计数错误。本研究专门研发的染色体测序核型分析方法相较传统的染色体核型分析方法还具有许多其它优势。首先,可快速分析母体核型,以作为NIPT检测结果的进一步解释。其它,作为一项分子检测技术,该方法具有可扩展性。再次,该方法具有很高的检测灵敏度,不仅可以检测母体X染色体的异常,甚至可检测低至5%的嵌合体。在少数情况下,这种方法也能检测常染色体核型的异常。鉴于这些优势,我们建议任何NIPT异常结果都需进行母体染色体核型的检测,以提高NIPT对性染色体检测结果的准确性。
Table 1. Detailed analysis and follow-up of NIPT results with unusually high or low ChrX z scores. 表1.X染色体NIPT 检测Z值异常高或异常低的样本的进一步详细分析 | |||||
NIPT sample no. NIPT样品编号: | NIPT ChrX z score NIPT X染色体z值 | Calculated NIPT fetal ChrX gain/loss NIPT计算的X染色体缺失/重复 | Fetal karyotypea 胎儿染色体核型a | Maternal WBC ChrX gain/loss by sequencing 母体白细胞测序所得的母体X染色体缺失/重复 | Maternal karyotype by G-bandingb 母体G显带染色体核型b |
1 | 51.55 | +79.31% | 46,XN | +103.93% | 47,XXX |
2 | -7.95 | -13.46% | 46,XN | -4.82% | 45,X[64]/46,XX[36] |
3 | -21.84 | -31.99% | 46,XN | -19.97% | 45,X[10]/46,XX[40] |
4 | 29.34% | +48.95% | 46,XN | +47.18% | 47,XXX[60]/46,XX[40] |
5 | -10.53% | -11.82% | 46,XN | -14.35% | 45,X[6]/46,XX[78] |
6 | 62.80 | +87.03% | 46,XN | +93.69% | 47,XXX |
a N, ChrX or ChrY. a N,X染色体或Y染色体 b Square brackets indicate the number of metaphases analyzed. b方括号表示分裂中期细胞计数 | |||||
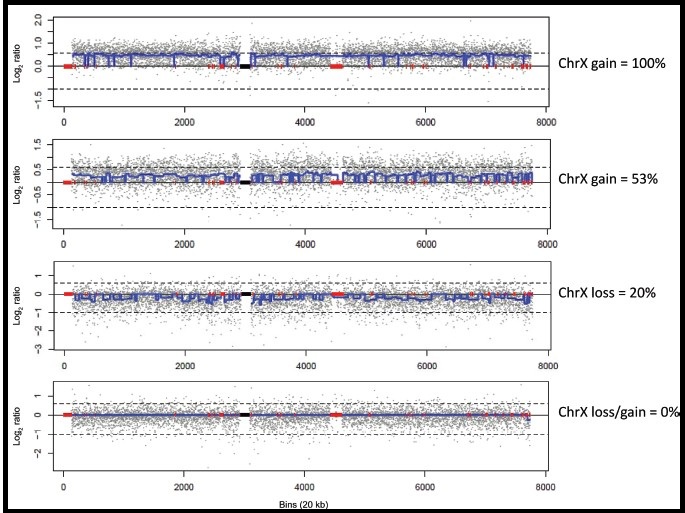
图4:测序检测来自NIPT的母体白细胞样本,分析母体X染色体异常或嵌合
母体白细胞测序显示NIPT样本的母体X染色体异常和嵌合
X染色体测序图,以标准化测序密度的log2值为Y轴,以20kb测序bin数为X轴。上虚线表示100%X染色体增加[log2(3/2)]。下虚线表示100% X染色体减少[log2(1/2)]。蓝色线代表样本log2值的平均值,红色方框表示序列重复区域,黑色方框表示着丝粒。以X染色体平均变化百分比为基础,上图显示,4名孕妇母体白细胞核型分别为X染色体93.7%重复、47.2%重复、20.0%缺失和无缺失/重复(正常46,XX核型)。
本研究结果也显示,确定母体染色体核型将大大降低NIPT SCAs检测的不符合率,并能够有效提高NIPT性染色体非整倍体检测的复合灵敏度和特异性。然而,对于母体X染色体低比例嵌合的情况NIPT仍然存在检测难度。
Table 2. Contribution of an abnormal ChrX maternal karyotype in a prospective study of 187 discordant SCAs.
表2:187例NIPT SCAs不符的前瞻性研究中母体X染色体的核型组成
| ||||
Clinical 临床 | NIPT findings NIPT结果 | NIPT ChrX gain NIPT X染色体重复 | NIPT ChrX loss NIPT X染色体缺失 | Total 总数 |
NIPT follow-up NIPT随访 | Abnormal NIPT for SCA, n NIPT SCA异常,n | 63 | 124 | 187 |
Normal maternal karyotype, n 母体染色体核型正常,n | 57 | 114 | 171 | |
Altered maternal karyotype, n 母体染色体核型异常,n | 6 | 10 | 16 | |
Maternal mosaicism rate 母体染色体嵌合比例 | 9.52%
| 8.06%
| 8.56%
| |
染色体核型异常的孕妇可孕育染色体正常的婴儿,也可孕育患有性染色体非整倍体疾病(SCAs)的婴儿(11,13)。虽然大多数情况下,母血浆中胎儿X染色体的真实情况会被异常的母体X染色体所掩盖,但当母体染色体存在异常时,对胎儿可能存在的潜在的性染色体非整倍体疾病(SCAs)也应提高警觉。极少数情况下,胎儿的SCA被母体的X嵌合而平衡,从而漏检。下面的假设证实了这种可能性。如果胎儿是XXX,母亲是10%的XO和90%的XX,如果胎儿的DNA片段比例约为10%,那么在不知道母体染色体核型的情况下,NIPT结果正常。如果NIPT正常,且知道母体染色体核型异常,需立即对孕妇进行核型确定,也可对胎儿进行羊水穿刺和染色体核型分析。如果有一种独立可靠的方法可以计算出基因组测序所得序列中的胎儿DNA片段(与X和Y染色体相关的除外),那么这种方法最终可以替代母体染色体测序法,可以更全面的检测母体X染色体异常或胎儿X染色体异常。
基于本研究中母体染色体嵌合是导致大部分NIPT SCAs检测不符原因,我们建议,不管临床应用的是基于鸟枪法的NIPT,还是基于单核苷酸多态性的NIPT,应对所有NIPT检测异常的样品进行母体染色体核型的检测。该建议可能引发伦理问题,特别是在先前尚未做过母体染色体核型分析以及并不知道母体染色体异常的情况下。鉴于这种可能性,需要对NIPT知情同意书进行修改,以阐明少数胎儿性染色体非整倍体(SCAs)是由母体染色体嵌合导致。另外,对这种情况的任何检测结果,应建议孕妇进行遗传咨询,以进一步讨论生育问题以及由于染色体异常造成的潜在健康问题。目前NIPT面临的最大挑战是胎盘嵌合可通过增加或减少胎儿相应染色体的有效DNA含量而引发假阳性与假阴性。虽然假阳性结果可通过羊水穿刺和染色体核型分析进行验证,从而避免对原本健康胎儿终止妊娠,但假阴性结果仍存在较多问题,特别是对怀孕期间无任何明显临床症状的性染色体非整倍体(SCAs)。
参考文献:
1. Kuliev A, Verlinsky Y. Meiotic and mitotic nondisjunction: lessons from preimplantation genetic diagnosis. Hum Reprod Update 2004;10:401-7.
2. Eiben B, Bartels I, B?hr-Porsch S, Borgmann S, Gatz G, Gellert G, et al. Cytogenetic analysis of 750 spontaneous abortions with the direct-preparation method of chorionic villi and its implications for studying genetic causes of pregnancy wastage. Am J Hum Genet 1990;47:656-63.
3. Stephenson MD, Awartani KA, Robinson WP. Cytogenetic analysis of miscarriages from couples with recurrent miscarriage: a case-control study. Hum Reprod 2002;17:446-51.
4. Simpson JL. Causes of fetal wastage. Clin Obstet Gynecol 2007;50:10-30.
5. Hassold T, Hunt P. To err (meiotically) is human: the genesis of human aneuploidy. Nat Rev Genet 2001;2:280-91.
6. Driscoll DA, Gross S. Prenatal screening for aneuploidy. N Engl J Med 2009;360:2556-62.
7. Raymond FL, Whittaker J, Jenkins L, Lench N, Chitty LS. Molecular prenatal diagnosis: the impact of modern technologies. Prenat Diagn 2010;30:674-81.
8. Natoli JL, Ackerman DL, McDermott S, Edwards JG. Prenatal diagnosis of Down syndrome: a systematic review of termination rates (1995-2011). Prenat Diagn 2012;32:142-53.
9. Benn P, Cuckle H, Pergament E. Non-invasive prenatal testing for aneuploidy: current status and future prospects. Ultrasound Obstet Gynecol 2013;42:15-33.
10. Luthardt, F. W. and Keitges, E. 2001. Chromosomal Syndromes and Genetic Disease. eLS.
11. Demaliaj E, Cerekja A, Piazze J. Sex Chromosome Aneuploidies. Aneuploidy in Health and Disease Ed Zuzana Storchova. In Tech; 2012:123-40.
12. Grati FR, Barlocco A, Grimi B, Milani S, Frascoli G, Di Meco AM, et al. Chromosome abnormalities investigated by non-invasive prenatal testing account for approximately 50% of fetal unbalances associated with relevant clinical phenotypes. Am J Med Genet 2010;152A:1434-42.
13. Bianchi DW, Platt LD, Goldberg JD, Abuhamad AZ, Sehnert AJ, Rava RP. Genome-wide fetal aneuploidy detection by maternal plasma DNA sequencing. Obstet Gynecol 2012;119:890-901.
14. Norton ME, Brar H, Weiss J, Karimi A, Laurent LC, Caughey AB, et al. Non-Invasive Chromosomal Evaluation (NICE) Study: results of a multicenter prospective cohort study for detection of fetal trisomy 21 and trisomy 18. Am J Obstet Gynecol 2012;207:137 e1-8.
15. Song Y, Liu C, Qi H, Zhang Y, Bian X, Liu J et al. Non invasive prenatal testing of fetal aneuploidies by massively parallel sequencing in a prospective Chinese population. Prenat Diagn 2013;33:700-6.
16. Choi H, Lau TK, Jiang FM, Chan MK, Zhang HY, Lo PS et al. Fetal aneuploidy screening by maternal plasma DNA sequencing: 'false positive' due to confined placental mosaicism. Prenat Diagn 2013;33:198-200.
17. Pan M, Li FT, Li Y, Jiang FM, Li DZ, Lau TK et al. Discordant results between fetal karyotyping and non-invasive prenatal testing by maternal plasma sequencing in a case of uniparental disomy 21 due to trisomic rescue. Prenat Diagn 2013;33:598-601.
18. Lau TK, Jiang FM, Stevenson RJ, Lo TK, Chan LW, Chan MK et al. Secondary findings from non-invasive prenatal testing for common fetal aneuploidies by whole genome sequencing as a clinical service. Prenat Diagn 2013; 33:602-8.
19. Hall AL, Drendel HM, Verbrugge JL, Reese AM, Schumacher KL, Griffith CB JL et al. Positive cell-free fetal DNA testing for trisomy 13 reveals confined placental mosaicism. Genet Med 2013; Mar 14. doi: 10.1038/gim.2013.26. [Epub ahead of print].
20. Canick JA, Palomaki GE, Kloza EM, Lambert-Messerlian GM, Haddow JE et al. The impact of maternal plasma DNA fetal fraction on next generation sequencing tests for common fetal aneuploidies. Prenat Diagn 2013;33:667-74.
21. Wang Y, Zhu J, Chen Y, Zhao X, Wu Y, Han X et al. Two cases of placental T21 mosaicism: challenging the detection limits of non-invasive prenatal testing. Prenat Diagn DOI: 10.1002/pd.4212.
22. Samango-Sprouse C, Banjevic M, Ryan A, Sigurjonsson S, Zimmermann B, Hill M et al. SNP-based non-invasive prenatal testing detects sex chromosome aneuploidies with high accuracy. Prenat Diagn 2013;33:643-9.
23. Mazloom AR, D?akula ?, Oeth P, Wang H, Jensen T, Tynan J et al. Noninvasive prenatal detection of sex chromosomal aneuploidies by sequencing circulating cellfree DNA from maternal plasma. Prenat Diagn 2013;33:591-7.
24. Skaletsky H, Kuroda-Kawaguchi T, Minx PJ, Cordum HS, Hillier L, Brown LG et al. The male-specific region of the human Y chromosome is a mosaic of discrete sequence classes. Nature 2003;423:825-37.
25. Osborne CM, Hardisty E, Devers P, Kaiser-Rogers K, Hayden MA, Goodnight W et al. Discordant noninvasive prenatal testing results in a patient subsequently diagnosed with metastatic disease. Prenat Diagn. 2013;33:609-11.
26. Russell LM, Strike P, Browne CE, Jacobs PA. X chromosome loss and aging. Cytogenet Genome Res 2007;116:181-5.
27. Liang D, Lv W, Wang H, Xu L, Liu J, Li H et al. Non-invasive prenatal testing of fetal whole chromosome aneuploidy by massively parallel sequencing. Prenat Diagn 2013;33:409-15.
28. Li H, Durbin R. Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics 2009;25:1754-60.
29. Tibshirani R, Wang P. Spatial smoothing and hot spot detection for CGH data using the fused lasso. Biostatistics 2008;9:18-29.
30. Moertel CA, Stupca PJ, Dewald GW. Pseudomosaicism, true mosaicism, and maternal cell contamination in amniotic fluid processed with in situ culture and robotic harvesting. Prenat Diag 1992;671-83.
31. Onalan G, Yilmaz Z, Durak T, Sahin FI, Zeyneloglu HB. Successful pregnancy with preimplantation genetic diagnosis in a woman with mosaic Turner syndrome. Fertil Steril 2011;95:1788.e1-3.
 妇产科在线APP下载
妇产科在线APP下载













